cfDNA甲基化組多模式分析早期檢測食管鱗狀細胞癌和癌前病變
食管癌(EC)是最常見的胃腸道惡性腫瘤之一,食管鱗狀細胞癌(ESCC)是EC的主要組織學亞型,約占新EC病例的88%,大多數發生在東亞和中亞。與晚期ESCC的預后極差相比,早期ESCC(如黏膜內ESCC)和前體病變(如上皮內瘤變,IEN)可以通過內鏡下整塊切除術實現近100%的五年疾病特異性生存率,從而無需系統性治療。因此檢測早期食管鱗狀細胞癌(ESCC)和癌前病變對于提高生存率至關重要。
液體活檢能夠檢測血漿中游離細胞DNA(cfDNA)內的循環腫瘤DNA(ctDNA),為非侵入性早期癌癥檢測提供了希望。然而關于液體活檢在ESCC診斷中應用的研究還很少。在最近對循環游離細胞基因組圖譜(CCGA)中cfDNA的多組學分析中,全基因組cfDNA甲基化成為癌癥檢測最有希望的信號,優于片段化標記和基因突變(如拷貝數變異,CNV)。然而,基于甲基化的ctDNA檢測方法仍存在一些挑戰。
中國醫學科學院北京協和醫學院劉芝華團隊、陳洪巖團隊和溫州醫科大學眼科醫院甌江實驗室蘇建忠團隊合作對來自230名非轉移性ESCC或癌前病變患者和230名來自多個中心的匹配健康對照(HC)的共460個cfDNA樣本進行了全基因組重亞硫酸鹽測序(WGBS)。并開發了一種擴展多模式分析(expanded multimodal analysis,EMMA)綜合方法,可以同時鑒定cfDNA WGBS數據中的cfDNA甲基化、拷貝數變異(CNV)和片段化標記。cfDNA甲基化標記是最早期和最敏感,可在70%的ESCC和50%的癌前病變中檢測到,并與分子亞型和腫瘤微環境相關。CNV和片段化標記表現出高特異性,但與晚期疾病有關。EMMA方法顯著提高了檢出率,將AUC(area under the curve)從0.90增加到0.99,并在驗證隊列中檢測到87%的ESCC和62%的癌前病變,特異性>95%。研究通過多模式分析方法分析了cfDNA-WGBS數據,揭示了cfDNA甲基化組的多模式分析對于早期檢測和監測ESCC分子特征的潛力。相關研究成果于2024年5月2日以“Multimodal analysis of cfDNA methylomes for early detecting esophageal squamous cell carcinoma and precancerous lesions”為題發表在《Nature Communications》(IF 16.6/Q1)期刊上。

cfDNA全基因組重亞硫酸鹽測序中的EMMA framework
為加強ESCC的早期檢測,研究人員基于“組織-cfDNA-組織”策略開發了EMMA framework方法(圖1a)。從配對的WGBS和原發性腫瘤的全基因組測序(WGS)數據中鑒定出ESCC的DMR和CNV,并從之前的隊列ESCC基因組和表觀基因組圖譜(ECGEA)中匹配了155例ESCC病例的鄰近癌旁組織。隨后分析了cfDNA WGBS數據中ESCC的DMR和CNV。基于腫瘤細胞的片段比正常細胞的片段更短,研究人員進一步計算了短cfDNA片段大小的比例作為cfDNA-WGBS數據中的片段大小比(FSR)。接下來使用包含150名ESCC患者和150名匹配的HC的數據集,采用隨機森林的機器學習方法,利用DMR、DMR與CNV、所有三個功能(DMR、CNV和FSR),在外部ESCC隊列和癌前隊列中獨立評估每種診斷模型的性能。此外將最佳DMR與成對ESCC組織樣品中基于多組學的整合分子亞型、腫瘤微環境(TME)、存活率和轉錄組學圖譜進行關聯分析。
研究設計了不同的隊列,包括來自中國醫學科學院癌癥醫院和中國北京協和醫學院的150名未經治療的ESCC或食管高度上皮內瘤變患者(HGIEN,即0期ESCC)(CHCAMS,發現/訓練隊列),來自上海胸科醫院的30名未經治療的ESCC患者(外部驗證隊列),來自CHCAMS的50名食管IEN患者(癌前驗證隊列)和230名HC,每位患者的年齡和性別在各自的隊列中相匹配(圖1b)。在進行任何醫療干預之前,研究人員從每位受試者(n=460)中位數收集2mL血漿。ESCC患者和對照組cfDNA濃度一致。WGBS用于評估每位受試者的cfDNA甲基化組,460個cfDNA樣品占參考基因組的約89%覆蓋率,平均深度為9.51X。
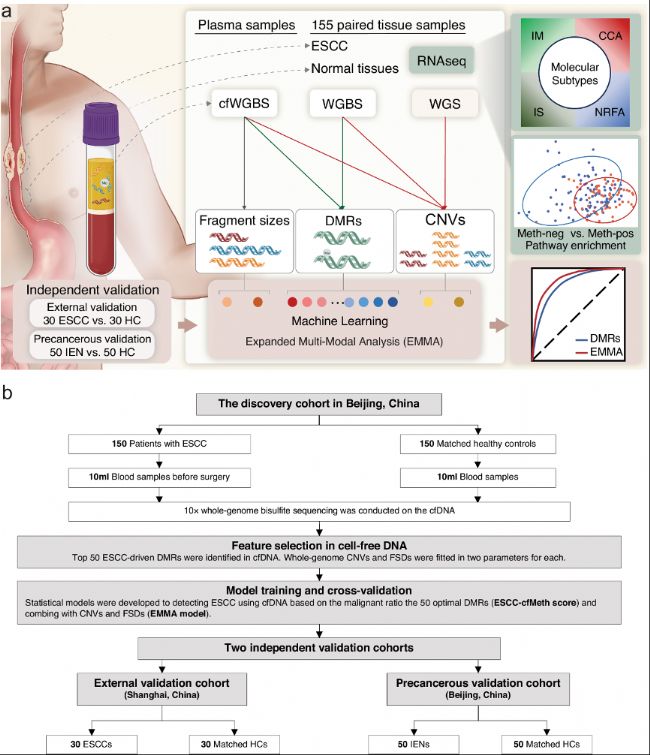
b. 該發現隊列包括150名ESCC或高級別上皮內瘤變患者和150名匹配的健康對照,以使用不同的cfDNA特征構建診斷模型。在外部ESCC隊列和癌前隊列中獨立評估每個診斷模型的性能。
ESCC:食管鱗狀細胞癌,IEN:上皮內瘤變,WGS:全基因組測序,WGBS:全基因組亞硫酸鹽測序,cfWGBS:cfDNA WGBS,RNAseq:RNA測序,HC:健康對照,CNV:拷貝數變異,DMR:差異甲基化區域,IM:免疫調節,CCA:細胞周期通路激活,IS:免疫抑制,NRFA:NRF2致癌激活。
cfDNA甲基化標記物的鑒定和性能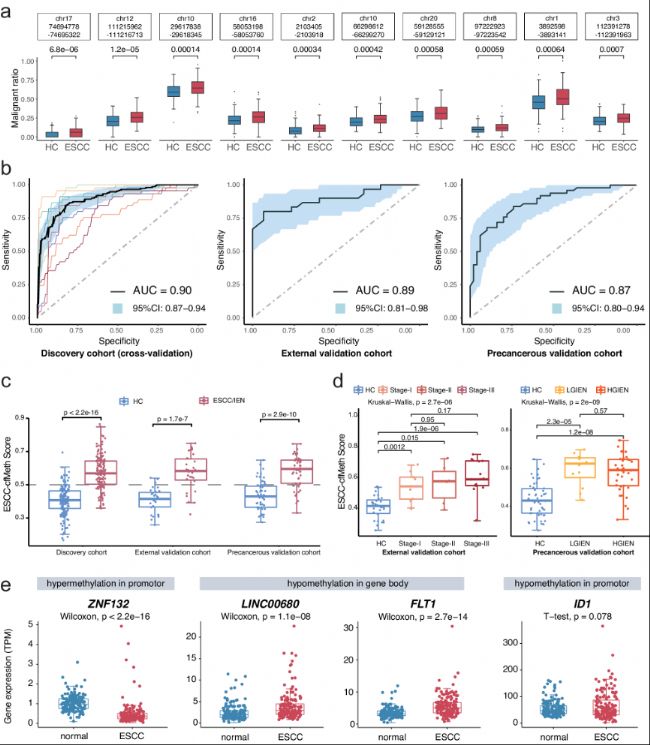
圖2:食管鱗狀細胞癌的游離DNA甲基化標志物及其性能檢測。
a. 在食管鱗狀細胞癌(ESCC)組織中發現的差異甲基化區域(DMRs)中,通過調整后的p值<0.05(雙側Wilcoxon檢驗)recall了650個DMRs,這些DMRs在發現隊列中的ESCC平均值(n=150)比健康對照組(n=150)更為顯著。圖中展示了前十個DMRs的惡性比例和p值。數據以最大值和最小值的中值表示。
b. ESCC-cfMeth評分的診斷性能在發現隊列(十倍交叉驗證,每種顏色的曲線表示一次交叉驗證)、外部驗證隊列和癌前驗證隊列中進行了評估。黑色曲線表示受試者ROC曲線,藍色區域表示95%置信區間(CI)。
c. 使用最佳50個標記的cfDNA惡性比例構建最終預測模型(ESCC-cfMeth評分)。與發現隊列和驗證隊列中的健康對照(HCs)相比,ESCC和上皮內瘤變(IEN)患者的ESCC-cfMeth評分顯著更高(雙側Mann-Whitney U檢驗,p<0.01)。數據以最大值和最小值的中值表示。
d. 與健康對照組相比,不同階段的ESCC(左側;n=30、9、6和 15,分別為各階段的樣本數)和IENs(右側;n=50、12和 38,分別為各階段的樣本數)的ESCC-cfMeth評分顯著提高。數據以最大值和最小值的中值表示。
e. 在ESCC組織和配對的相鄰癌旁組織(n=155)之間的基因表達比較中,發現50個最佳DMRs中的功能基因表達存在差異(使用雙側Wilcoxon檢驗或t檢驗,ZNF132的p值<2.2×10^-16,LINC00680、FLT1和ID1的p值分別為1.1×10^-8、2.7×10^-14和0.078)。
cfDNA中鑒定CNV用于ESCC檢測
圖3:cfDNA全基因組亞硫酸鹽測序數據中的拷貝數變異事件的recalling和分析
a. 基于全基因組重亞硫酸鹽測序(WGBS)方法,從組織和cfDNA中recalling WGBS數據中的重復拷貝數變異(CNV)。
b. 以ECGEA隊列中的002號患者為例,在全基因組測序數據中鑒定出chr.3、chr. 5的擴增和chr. 3、4、9、10、11、13、16、18、21的缺失,并在成對的WGBS數據中recall。
c. 150例食管鱗狀細胞癌(ESCC)患者中,153個區域的CNV事件發生率顯著高于150例健康對照組(HCs)。擴增(紅色)和缺失(藍色)用ESCC與HCs差異的相應調整p值(錯誤發現率,FDR)顯示(雙側t檢驗;ns,灰色;FDR<0.05,黃色;FDR<0.01,橙色;FDR<0.001,深紅色)。
d. ESCC和上皮內瘤變(IEN)患者的CNV陽性率顯著高于發現隊列和驗證隊列中的HCs,并且與分期和分級呈正相關。
ESCC:食管鱗狀細胞癌,IEN:上皮內瘤變,LGIEN:低級別IEN,HGIEN:高級別IEN,HC:健康對照,WGS:全基因組測序,WGBS:全基因組重亞硫酸鹽測序,GMM:高斯混合模型,HMM:隱馬爾可夫模型,CNV:拷貝數變異,AMPL:擴增,DELE:缺失,ns:無顯著性,G級。
cfDNA-WGBS數據中片段大小的檢測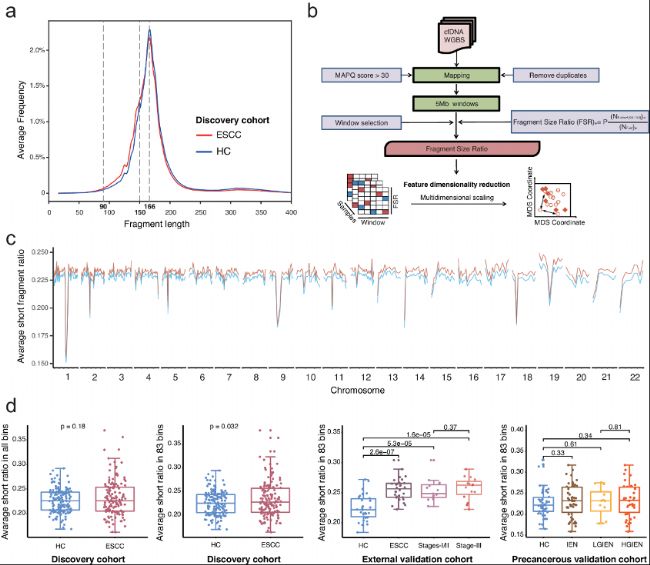
圖4:分析全基因組重亞硫酸鹽測序數據中的cfDNA片段大小。
a. 在發現隊列中分析cfDNA片段大小。食管鱗狀細胞癌(ESCC)患者和健康對照組的cfDNA peaks均為166 bp。但在ESCC中鑒定出更多的短片段(90–150bp)。
b. 以全基因組重亞硫酸鹽測序數據中的片段大小比(FSR)計算cfDNA的短片段比例。
c. 人類基因組分為5Mb bins,共產生1082(541個bins×2)個FSR。
d. 在發現隊列中,ESCC患者和HCs之間所有bins的平均FSR均無顯著差異。但83個bins中ESCC患者的FSR顯著高于發現隊列中HCs。發現隊列和外部驗證隊列中的ESCC患者中83個選定BIN的平均FSR顯著高于癌前驗證隊列中的上皮內瘤變患者(雙側Mann–Whitney U檢驗,p < 0.05)。數據以具有最大值和最小值的中值表示。
聯合EMMA模型準確檢測ESCC和癌前病變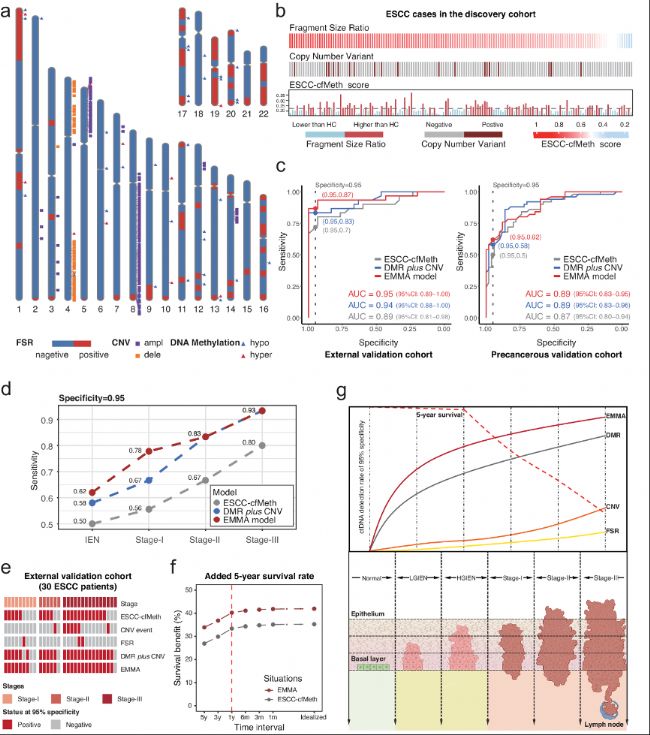
b. 在發現隊列的食管鱗狀細胞癌(ESCC)患者中發現了這三個標記之間的互補性。每個條形圖表示ESCC患者中每個標記的狀態。
c. 在外部驗證隊列和癌前驗證隊列中評估ESCC-cfMeth評分、DMR加CNV模型、和EMMA模型的診斷性能。
d. ESCC-cfMeth評分、DMR加CNV模型、和EMMA模型對上皮內瘤變(IEN)I、II和III期ESCC的檢出率。
e. 在外部驗證隊列中,三個標記的互補性提高了組合模型的性能。
f. 根據不同的實驗間隔,從5年到連續測試(理想化),評估EMMA模型和ESCC-cfMeth模型的潛在生存效益。
g. 不同cfDNA標記和組合模型的檢出率以及不同階段的5年生存率示意圖。
cfDNA甲基化標記的生物學意義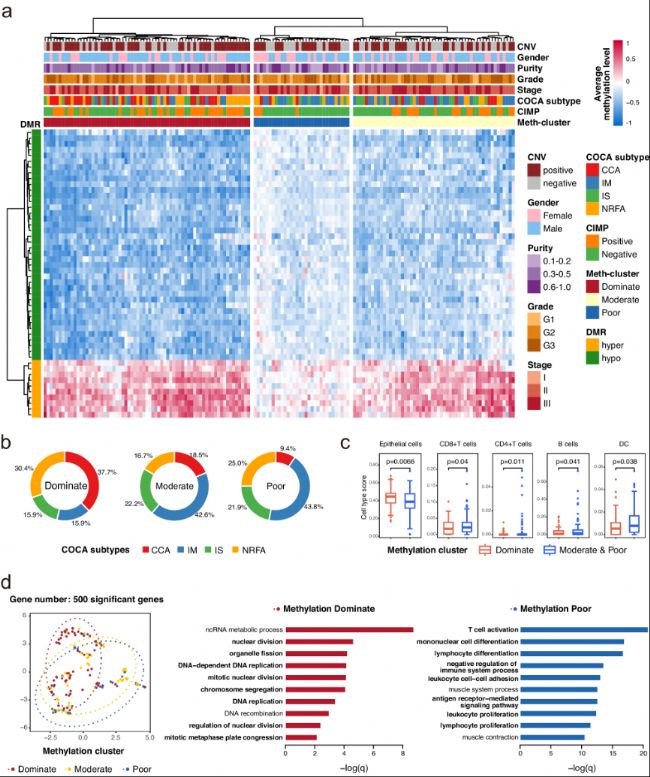
b. ESCC分子亞型在甲基化顯著組(methylation-dominate)(n=69)、甲基化中度組(methylation-moderate)(n=54)和甲基化缺失組(methylation-poor)(n=32)中的比例。
c. 通過雙側t檢驗對甲基化顯著組(n=69)和甲基化中度組/甲基化缺失組(n=86)比較分析腫瘤微環境中的細胞組分。(p=6.6×10-3、0.04、0.01、0.04和0.04分別用于上皮細胞、CD4+T細胞、CD8+T細胞、B細胞和樹突狀細胞)。
d. 這些通路在甲基化顯著組和甲基化缺失組中富集。
研究小結:
本研究分析了循環游離DNA(cfDNA)中鑒定出的最佳甲基化標記的生物學意義。研究首先從大量配對的食管鱗狀細胞癌(ESCC)組織樣本中獲得的全基因組亞硫酸鹽測序(WGBS)數據中鑒定了差異甲基化區域(DMRs),并通過與多種細胞類型的比較驗證其特異性。在這些DMRs中發現了與ESCC相關且表達顯著不同的已知功能基因。隨后根據最佳DMRs的甲基化水平將ESCC患者分為三組。甲基化顯著組表現出更高的CCA亞型和上皮細胞組分患病率,這也與更好的存活率相關。相比之下,腫瘤微環境(TME)的分析和差異表達顯示,免疫細胞的升高和免疫過程的增強可能會減少與ESCC相關甲基化標記信號。甲基化中等組和甲基化缺失組屬于IM亞型的ESCC患者比例較高。這一亞型對免疫療法的靈敏度高于其他ESCC亞型,表明cfDNA檢測在預測和監測免疫療法反應方面的潛力。總體而言,本研究為cfDNA甲基化標記的生物學和臨床意義提供了全面的見解,并提供了一種用于動態監測ESCC分子特征的非侵入性工具。
同時本研究有幾個局限性:
a)盡管分析了1000多個全基因組數據集,但本研究驗證隊列相對較小。
b)這些多模式標記的潛在功能仍然未知,尤其是在癌前病變中。
c)本研究是以成本效益高且節省樣本的方式從WGBS數據中獲得了拷貝數變異(CNVs)和片段化標記,但在WGBS中的亞硫酸鹽轉化過程中,一些cfDNA片段可能會受損,與直接從cfDNA WGS數據相比,可能會削弱CNVs和片段化標記的信號。
d)由于在WGBS數據中突變calling的不可靠性,本多模式模型中未納入突變。使用其他方法準確鑒定TP53等基因突變可能會進一步提高ESCC的檢出率。
e)ESCC-cfDNA和EMMA模型的生存效益和預后意義需要在大規模真實世界隊列中進一步驗證。
總之,本研究通過多模式方法對液體活檢中的cfDNA甲基化、CNV和片段化標記進行了全面分析,以實現ESCC的超早期檢測。通過多模式方法分析cfDNA WGBS數據鑒定了幾種分期特異性標記及其互補性。研究不僅顯著提高了ESCC的非侵入性檢測能力,而且還具有動態分子監測和治療指導的潛力。
參考文獻:
Liu J, Dai L, Wang Q, Li C, Liu Z, Gong T, Xu H, Jia Z, Sun W, Wang X, Lu M, Shang T, Zhao N, Cai J, Li Z, Chen H, Su J, Liu Z. Multimodal analysis of cfDNA methylomes for early detecting esophageal squamous cell carcinoma and precancerous lesions. Nat Commun. 2024 May 2;15(1):3700. pii: 10.1038/s41467-024-47886-1. doi: 10.1038/s41467-024-47886-1. PubMed PMID: 38697989.
液體活檢能夠檢測血漿中游離細胞DNA(cfDNA)內的循環腫瘤DNA(ctDNA),為非侵入性早期癌癥檢測提供了希望。然而關于液體活檢在ESCC診斷中應用的研究還很少。在最近對循環游離細胞基因組圖譜(CCGA)中cfDNA的多組學分析中,全基因組cfDNA甲基化成為癌癥檢測最有希望的信號,優于片段化標記和基因突變(如拷貝數變異,CNV)。然而,基于甲基化的ctDNA檢測方法仍存在一些挑戰。
中國醫學科學院北京協和醫學院劉芝華團隊、陳洪巖團隊和溫州醫科大學眼科醫院甌江實驗室蘇建忠團隊合作對來自230名非轉移性ESCC或癌前病變患者和230名來自多個中心的匹配健康對照(HC)的共460個cfDNA樣本進行了全基因組重亞硫酸鹽測序(WGBS)。并開發了一種擴展多模式分析(expanded multimodal analysis,EMMA)綜合方法,可以同時鑒定cfDNA WGBS數據中的cfDNA甲基化、拷貝數變異(CNV)和片段化標記。cfDNA甲基化標記是最早期和最敏感,可在70%的ESCC和50%的癌前病變中檢測到,并與分子亞型和腫瘤微環境相關。CNV和片段化標記表現出高特異性,但與晚期疾病有關。EMMA方法顯著提高了檢出率,將AUC(area under the curve)從0.90增加到0.99,并在驗證隊列中檢測到87%的ESCC和62%的癌前病變,特異性>95%。研究通過多模式分析方法分析了cfDNA-WGBS數據,揭示了cfDNA甲基化組的多模式分析對于早期檢測和監測ESCC分子特征的潛力。相關研究成果于2024年5月2日以“Multimodal analysis of cfDNA methylomes for early detecting esophageal squamous cell carcinoma and precancerous lesions”為題發表在《Nature Communications》(IF 16.6/Q1)期刊上。

cfDNA全基因組重亞硫酸鹽測序中的EMMA framework
為加強ESCC的早期檢測,研究人員基于“組織-cfDNA-組織”策略開發了EMMA framework方法(圖1a)。從配對的WGBS和原發性腫瘤的全基因組測序(WGS)數據中鑒定出ESCC的DMR和CNV,并從之前的隊列ESCC基因組和表觀基因組圖譜(ECGEA)中匹配了155例ESCC病例的鄰近癌旁組織。隨后分析了cfDNA WGBS數據中ESCC的DMR和CNV。基于腫瘤細胞的片段比正常細胞的片段更短,研究人員進一步計算了短cfDNA片段大小的比例作為cfDNA-WGBS數據中的片段大小比(FSR)。接下來使用包含150名ESCC患者和150名匹配的HC的數據集,采用隨機森林的機器學習方法,利用DMR、DMR與CNV、所有三個功能(DMR、CNV和FSR),在外部ESCC隊列和癌前隊列中獨立評估每種診斷模型的性能。此外將最佳DMR與成對ESCC組織樣品中基于多組學的整合分子亞型、腫瘤微環境(TME)、存活率和轉錄組學圖譜進行關聯分析。
研究設計了不同的隊列,包括來自中國醫學科學院癌癥醫院和中國北京協和醫學院的150名未經治療的ESCC或食管高度上皮內瘤變患者(HGIEN,即0期ESCC)(CHCAMS,發現/訓練隊列),來自上海胸科醫院的30名未經治療的ESCC患者(外部驗證隊列),來自CHCAMS的50名食管IEN患者(癌前驗證隊列)和230名HC,每位患者的年齡和性別在各自的隊列中相匹配(圖1b)。在進行任何醫療干預之前,研究人員從每位受試者(n=460)中位數收集2mL血漿。ESCC患者和對照組cfDNA濃度一致。WGBS用于評估每位受試者的cfDNA甲基化組,460個cfDNA樣品占參考基因組的約89%覆蓋率,平均深度為9.51X。

圖1:研究設計和患者入組。
a. 使用機器學習開發了EMMA多模式分析方法,以加強血漿樣品cfDNA中ctDNA的檢測。該方法通過全面分析cfDNA全基因組亞硫酸鹽測序(cfWGBS)數據中癌癥來源的差異甲基化區域(DMR)、拷貝數變異(CNV)和片段化標記。最初從155例食管鱗狀細胞癌(ESCC)患者的原發性腫瘤和匹配的相鄰癌旁組織的配對WGBS數據和WGS數據中鑒定出癌癥來源的DMR和CNV。隨后在cfWGBS數據中分析了ESCC來源的DMR和CNV,并進一步利用短cfDNA片段大小比例來訓練發現隊列中的診斷模型。在外部ESCC隊列和癌前隊列中獨立評估了每種診斷模型的性能。為了揭示這些最佳DMR的生物學意義,研究人員將它們與成對ESCC組織樣品中基于多組學的分子亞型和轉錄組學譜相關聯。b. 該發現隊列包括150名ESCC或高級別上皮內瘤變患者和150名匹配的健康對照,以使用不同的cfDNA特征構建診斷模型。在外部ESCC隊列和癌前隊列中獨立評估每個診斷模型的性能。
ESCC:食管鱗狀細胞癌,IEN:上皮內瘤變,WGS:全基因組測序,WGBS:全基因組亞硫酸鹽測序,cfWGBS:cfDNA WGBS,RNAseq:RNA測序,HC:健康對照,CNV:拷貝數變異,DMR:差異甲基化區域,IM:免疫調節,CCA:細胞周期通路激活,IS:免疫抑制,NRFA:NRF2致癌激活。
cfDNA甲基化標記物的鑒定和性能

圖2:食管鱗狀細胞癌的游離DNA甲基化標志物及其性能檢測。
b. ESCC-cfMeth評分的診斷性能在發現隊列(十倍交叉驗證,每種顏色的曲線表示一次交叉驗證)、外部驗證隊列和癌前驗證隊列中進行了評估。黑色曲線表示受試者ROC曲線,藍色區域表示95%置信區間(CI)。
c. 使用最佳50個標記的cfDNA惡性比例構建最終預測模型(ESCC-cfMeth評分)。與發現隊列和驗證隊列中的健康對照(HCs)相比,ESCC和上皮內瘤變(IEN)患者的ESCC-cfMeth評分顯著更高(雙側Mann-Whitney U檢驗,p<0.01)。數據以最大值和最小值的中值表示。
d. 與健康對照組相比,不同階段的ESCC(左側;n=30、9、6和 15,分別為各階段的樣本數)和IENs(右側;n=50、12和 38,分別為各階段的樣本數)的ESCC-cfMeth評分顯著提高。數據以最大值和最小值的中值表示。
e. 在ESCC組織和配對的相鄰癌旁組織(n=155)之間的基因表達比較中,發現50個最佳DMRs中的功能基因表達存在差異(使用雙側Wilcoxon檢驗或t檢驗,ZNF132的p值<2.2×10^-16,LINC00680、FLT1和ID1的p值分別為1.1×10^-8、2.7×10^-14和0.078)。
cfDNA中鑒定CNV用于ESCC檢測

圖3:cfDNA全基因組亞硫酸鹽測序數據中的拷貝數變異事件的recalling和分析
b. 以ECGEA隊列中的002號患者為例,在全基因組測序數據中鑒定出chr.3、chr. 5的擴增和chr. 3、4、9、10、11、13、16、18、21的缺失,并在成對的WGBS數據中recall。
c. 150例食管鱗狀細胞癌(ESCC)患者中,153個區域的CNV事件發生率顯著高于150例健康對照組(HCs)。擴增(紅色)和缺失(藍色)用ESCC與HCs差異的相應調整p值(錯誤發現率,FDR)顯示(雙側t檢驗;ns,灰色;FDR<0.05,黃色;FDR<0.01,橙色;FDR<0.001,深紅色)。
d. ESCC和上皮內瘤變(IEN)患者的CNV陽性率顯著高于發現隊列和驗證隊列中的HCs,并且與分期和分級呈正相關。
ESCC:食管鱗狀細胞癌,IEN:上皮內瘤變,LGIEN:低級別IEN,HGIEN:高級別IEN,HC:健康對照,WGS:全基因組測序,WGBS:全基因組重亞硫酸鹽測序,GMM:高斯混合模型,HMM:隱馬爾可夫模型,CNV:拷貝數變異,AMPL:擴增,DELE:缺失,ns:無顯著性,G級。
cfDNA-WGBS數據中片段大小的檢測

圖4:分析全基因組重亞硫酸鹽測序數據中的cfDNA片段大小。
b. 以全基因組重亞硫酸鹽測序數據中的片段大小比(FSR)計算cfDNA的短片段比例。
c. 人類基因組分為5Mb bins,共產生1082(541個bins×2)個FSR。
d. 在發現隊列中,ESCC患者和HCs之間所有bins的平均FSR均無顯著差異。但83個bins中ESCC患者的FSR顯著高于發現隊列中HCs。發現隊列和外部驗證隊列中的ESCC患者中83個選定BIN的平均FSR顯著高于癌前驗證隊列中的上皮內瘤變患者(雙側Mann–Whitney U檢驗,p < 0.05)。數據以具有最大值和最小值的中值表示。
聯合EMMA模型準確檢測ESCC和癌前病變

圖5:三種cfDNA特征的互補性和組合模型的性能。
a. 人類基因組中cfDNA甲基化標記、拷貝數變異和片段化標記分布。b. 在發現隊列的食管鱗狀細胞癌(ESCC)患者中發現了這三個標記之間的互補性。每個條形圖表示ESCC患者中每個標記的狀態。
c. 在外部驗證隊列和癌前驗證隊列中評估ESCC-cfMeth評分、DMR加CNV模型、和EMMA模型的診斷性能。
d. ESCC-cfMeth評分、DMR加CNV模型、和EMMA模型對上皮內瘤變(IEN)I、II和III期ESCC的檢出率。
e. 在外部驗證隊列中,三個標記的互補性提高了組合模型的性能。
f. 根據不同的實驗間隔,從5年到連續測試(理想化),評估EMMA模型和ESCC-cfMeth模型的潛在生存效益。
g. 不同cfDNA標記和組合模型的檢出率以及不同階段的5年生存率示意圖。
cfDNA甲基化標記的生物學意義

圖6:DNA甲基化標記在食管鱗狀細胞癌cfDNA中的生物學意義
a. 根據ESCC-cfMeth評分中50個最佳DNA甲基化標記的平均甲基化水平,將ECGEA隊列中的155例食管鱗狀細胞癌(ESCC)患者分為三組。b. ESCC分子亞型在甲基化顯著組(methylation-dominate)(n=69)、甲基化中度組(methylation-moderate)(n=54)和甲基化缺失組(methylation-poor)(n=32)中的比例。
c. 通過雙側t檢驗對甲基化顯著組(n=69)和甲基化中度組/甲基化缺失組(n=86)比較分析腫瘤微環境中的細胞組分。(p=6.6×10-3、0.04、0.01、0.04和0.04分別用于上皮細胞、CD4+T細胞、CD8+T細胞、B細胞和樹突狀細胞)。
d. 這些通路在甲基化顯著組和甲基化缺失組中富集。
研究小結:
本研究分析了循環游離DNA(cfDNA)中鑒定出的最佳甲基化標記的生物學意義。研究首先從大量配對的食管鱗狀細胞癌(ESCC)組織樣本中獲得的全基因組亞硫酸鹽測序(WGBS)數據中鑒定了差異甲基化區域(DMRs),并通過與多種細胞類型的比較驗證其特異性。在這些DMRs中發現了與ESCC相關且表達顯著不同的已知功能基因。隨后根據最佳DMRs的甲基化水平將ESCC患者分為三組。甲基化顯著組表現出更高的CCA亞型和上皮細胞組分患病率,這也與更好的存活率相關。相比之下,腫瘤微環境(TME)的分析和差異表達顯示,免疫細胞的升高和免疫過程的增強可能會減少與ESCC相關甲基化標記信號。甲基化中等組和甲基化缺失組屬于IM亞型的ESCC患者比例較高。這一亞型對免疫療法的靈敏度高于其他ESCC亞型,表明cfDNA檢測在預測和監測免疫療法反應方面的潛力。總體而言,本研究為cfDNA甲基化標記的生物學和臨床意義提供了全面的見解,并提供了一種用于動態監測ESCC分子特征的非侵入性工具。
同時本研究有幾個局限性:
a)盡管分析了1000多個全基因組數據集,但本研究驗證隊列相對較小。
b)這些多模式標記的潛在功能仍然未知,尤其是在癌前病變中。
c)本研究是以成本效益高且節省樣本的方式從WGBS數據中獲得了拷貝數變異(CNVs)和片段化標記,但在WGBS中的亞硫酸鹽轉化過程中,一些cfDNA片段可能會受損,與直接從cfDNA WGS數據相比,可能會削弱CNVs和片段化標記的信號。
d)由于在WGBS數據中突變calling的不可靠性,本多模式模型中未納入突變。使用其他方法準確鑒定TP53等基因突變可能會進一步提高ESCC的檢出率。
e)ESCC-cfDNA和EMMA模型的生存效益和預后意義需要在大規模真實世界隊列中進一步驗證。
總之,本研究通過多模式方法對液體活檢中的cfDNA甲基化、CNV和片段化標記進行了全面分析,以實現ESCC的超早期檢測。通過多模式方法分析cfDNA WGBS數據鑒定了幾種分期特異性標記及其互補性。研究不僅顯著提高了ESCC的非侵入性檢測能力,而且還具有動態分子監測和治療指導的潛力。
參考文獻:
Liu J, Dai L, Wang Q, Li C, Liu Z, Gong T, Xu H, Jia Z, Sun W, Wang X, Lu M, Shang T, Zhao N, Cai J, Li Z, Chen H, Su J, Liu Z. Multimodal analysis of cfDNA methylomes for early detecting esophageal squamous cell carcinoma and precancerous lesions. Nat Commun. 2024 May 2;15(1):3700. pii: 10.1038/s41467-024-47886-1. doi: 10.1038/s41467-024-47886-1. PubMed PMID: 38697989.
標簽:
DNA甲基化
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com