cfMeDIP-seq揭示cfDNA甲基化高效區分原發性和轉移性前列腺
大家好,這里是專注表觀組學十余年,領跑多組學科研服務的易基因。
前列腺癌(Prostate cancer,PCa)是男性中第二常見的惡性腫瘤,也是全球癌癥相關死亡的第三大原因。雖然大多數原發性前列腺癌可以治愈,但轉移性前列腺癌患者的5年生存率仍低至30%。大多數患者很快就會發展成轉移性去勢抵抗性前列腺癌(metastatic castration-resistant prostate cancer,mCRPC),最終導致死亡。因此轉移性前列腺癌仍是一個主要的臨床挑戰,轉移性病變高度異質性且難以活檢,而液體活檢提供了深入了解潛在生物學的機會。循環腫瘤DNA(ctDNA)在液體活檢中的分析已顯示出作為微創且準確的疾病監測工具潛力。在mCRPC中,ctDNA已被證明可以反映腫瘤或轉移性病變的基因組特征、甲基化狀態等表觀遺傳特征可以反映腫瘤負擔和亞型。但在大規模臨床隊列中仍然缺乏mCRPC的全基因組cfDNA甲基化研究。最近,細胞游離甲基化DNA免疫沉淀與下一代測序(cfMeDIP-seq)的發展為分析cfDNA甲基化組提供了一種有效的方法。這種方法可以從微量cfDNA中敏感地檢測ctDNA,并且與全基因組亞硫酸鹽測序方法相比更具成本效益。
近日,廈門大學健康醫療大數據國家研究院吉國力教授團隊和加拿大多倫多大學瑪格麗特公主癌癥中心何厚勝(Housheng Hansen He)教授團隊等合作在Nature子刊《nature communications》發表題為“The cell-free DNA methylome captures distinctions between localized and metastatic prostate tumors”的科研成果,研究利用cfMeDIP-seq技術分析了60個原發和175個轉移性前列腺癌樣本的細胞游離DNA(cfDNA)甲基化組,全基因組甲基化組高效捕獲了反映疾病異質性變異。進一步研究表明,cfDNA甲基化組可以以高準確率區分不同的疾病狀態,突出其作為疾病監測和預后的微創策略潛力。

標題:The cell-free DNA methylome captures distinctions between localized and metastatic prostate tumors(cfDNA甲基化組捕獲區分原發和轉移性前列腺癌)
期刊:nature communications
影響因子: IF 14.7 / 1區
技術平臺:cfMeDIP-seq
作者利用高度敏感的富集型測序技術cfMeDIP-seq,分別對4個研究隊列中60名原發前列腺癌患者和175名轉移性前列腺癌患者的cfDNA甲基化組進行了分析。分析結果揭示cfDNA甲基化組可以捕獲腫瘤變化。在轉移性樣本中觀察到全基因組高甲基化,同時著絲粒周圍區域出現低甲基化。糖皮質激素受體(glucocorticoid receptor,GR)基因NR3C1的啟動子區域高甲基化與免疫信號減少有關。cfDNA甲基化組反映了臨床結果,并且可以以98.9%高精度準確率預測區分不同的疾病類型(轉移性和原發樣本),表現出了強大的疾病監測和預測的微創策略潛力。最后,研究從數據中預測拷貝數變化的能力,為有限的生物樣本提供了聯合遺傳和表觀遺傳分析的機會。
研究設計
樣本收集:4個研究隊列(原發性腫瘤患者來自CPC-Gene,轉移性前列腺癌來自VPC、Barrier、WCDT)。分析方法:使用高靈敏度的基于富集測序技術cfMeDIP-seq,分析了60個局部前列腺癌和175個轉移性前列腺癌患者的血漿DNA甲基化組,將所有血漿樣本中的甲基化DNA片段沉淀并進行配對末端DNA測序。
結果圖形
(1)對原發和轉移性前列腺癌(PCa)的血漿游離DNA甲基化組進行全基因組范圍分析。
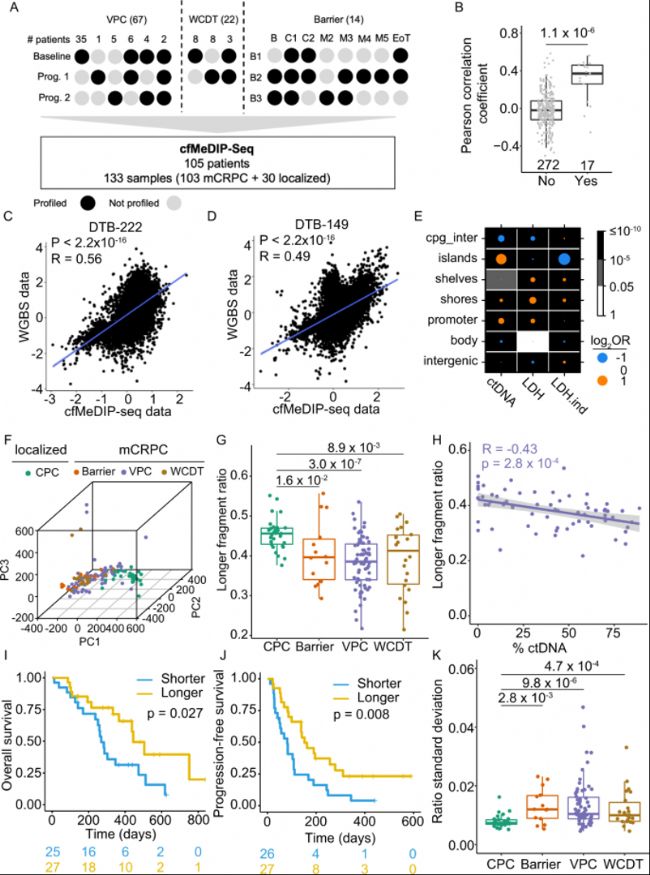
圖1:cfMeDIP數據捕獲原發與轉移性去勢抵抗性前列腺癌(mCRPC)患者血漿樣本中cfDNA甲基化和片段化變化。
A. 三個mCRPC隊列的抽樣示意圖。括號中數字表示給定隊列中的樣本總數。
B-D. WCDT隊列,組織WGBS和cfMeDIP數據的樣本配對相關性,比較匹配樣本與非匹配樣本的差異。
E. %ctDNA、LDH以及與不依賴%ctDNA的LDH解釋的不同基因組位點富集情況。
F. 前10,000個變異區間的主成分分析(PCA)對四個隊列樣本進行三維表示。
G. 各隊列中較長cfDNA片段比例。
H. VPC隊列中mCRPC樣本的較長片段比例與ctDNA比例(%ctDNA)的皮爾遜相關性。
I-J. 片段大小與總生存期(OS)和無進展生存期(PFS)的相關性。
K. 樣本內片段比例標準差分布。
(2)cfDNA甲基化分析顯示轉移性前列腺癌樣本中廣泛高甲基化和著絲粒周圍區域低甲基化
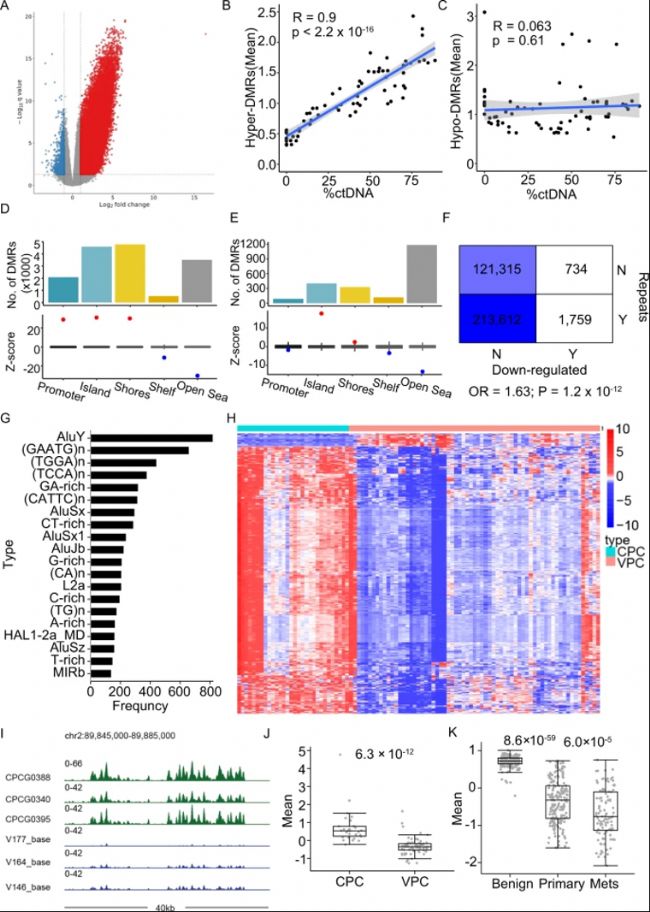
B-C. 高甲基化(Hyper)和低甲基化(Hypo)DMRs平均甲基化水平與%ctDNA之間的皮爾遜相關性。
D-E. 高甲基化和低甲基化DMRs的基因組分布。
F. 與低甲基化峰重疊的重復類型分布的列聯表。
G. 與下調峰重疊的重復類型頻率。
H. 位于著絲粒周圍1 Mb區域內的差異性甲基化峰。
I. 染色體2中著絲粒附近區域信號分布示例。
J-K. 分別展示了cfMeDIP-seq和WGBS數據中H中顯示的差異峰的平均甲基化水平。
(3)NR3C1啟動子甲基化水平與不同疾病結局相關
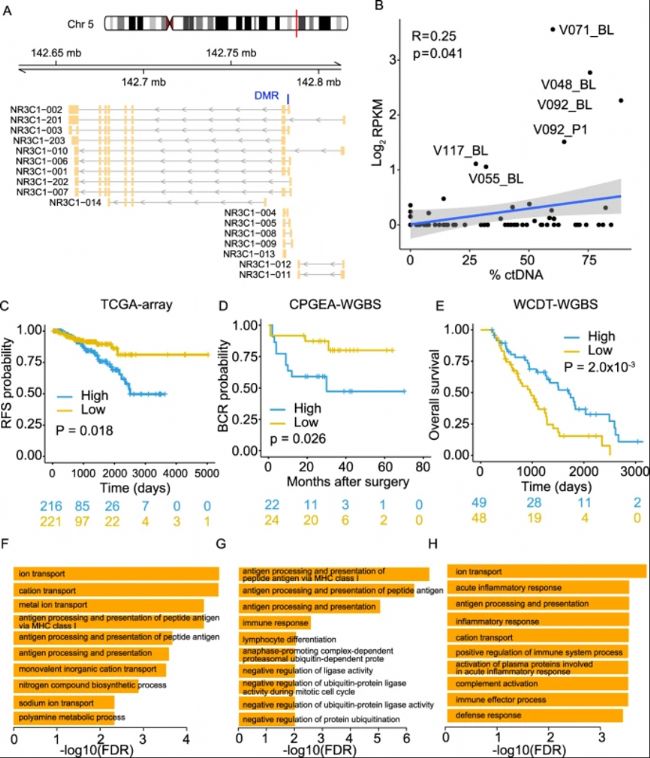
B. VPC隊列中GR位點甲基化水平與%ctDNA之間的皮爾遜相關性。
C-E. 癌癥基因組圖譜(TCGA)、中國前列腺癌基因組和表觀基因組圖譜(CPGEA)和WCDT隊列中GR位點甲基化與疾病結果之間的相關性。
F-H. TCGA(F)、CPGEA(G)和WCDT(H)隊列中高GR-DMR甲基化組中下調基因的生物學過程(BP)的基因本體(GO)富集分析。
(4)cfDNA甲基化組可高精度區分轉移性和原發性樣本
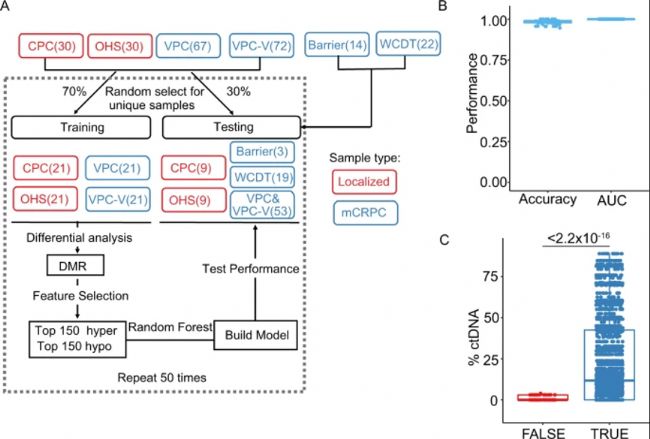
B. 測試數據集上,基于甲基化譜預測因子的預測準確性和接收者操作特征曲線下面積(AUROC)。VPC-V驗證樣本來自VPC隊列,OHS為安大略省健康研究隊列,DMR為差異甲基化區域。
C. 對于轉移樣本,正確或錯誤分類的樣本中%ctDNA分布。
(5)游離DNA甲基化組可以預測大規模遺傳變異
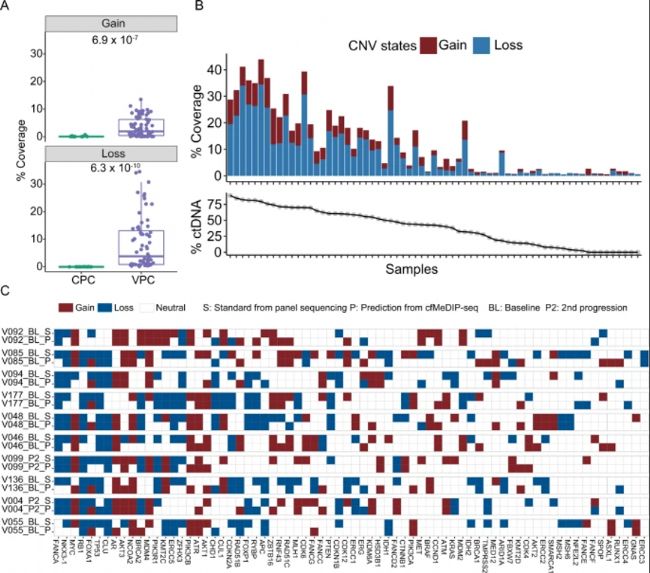
圖5:使用cfDNA甲基化組預測樣本拷貝數變異(CNA)。
A. 預測來自CPC隊列的原發樣本和VPC隊列的mCRPC樣本的CNA覆蓋度。
B. VPC隊列中mCRPC樣本預測的CNA覆蓋度與%ctDNA之間的相關性。
C. 比較先前panel測序獲得的標準狀態和cfMeDIP-seq數據預測結果的基因CNA,針對CNA覆蓋度最高的前10個樣本。
參考文獻:
Chen S, et al. The cell-free DNA methylome captures distinctions between localized and metastatic prostate tumors. Nat Commun. 2022 Oct 29;13(1):6467. pii: 10.1038/s41467-022-34012-2. doi: 10.1038/s41467-022-34012-2. PubMed PMID: 36309516.
前列腺癌(Prostate cancer,PCa)是男性中第二常見的惡性腫瘤,也是全球癌癥相關死亡的第三大原因。雖然大多數原發性前列腺癌可以治愈,但轉移性前列腺癌患者的5年生存率仍低至30%。大多數患者很快就會發展成轉移性去勢抵抗性前列腺癌(metastatic castration-resistant prostate cancer,mCRPC),最終導致死亡。因此轉移性前列腺癌仍是一個主要的臨床挑戰,轉移性病變高度異質性且難以活檢,而液體活檢提供了深入了解潛在生物學的機會。循環腫瘤DNA(ctDNA)在液體活檢中的分析已顯示出作為微創且準確的疾病監測工具潛力。在mCRPC中,ctDNA已被證明可以反映腫瘤或轉移性病變的基因組特征、甲基化狀態等表觀遺傳特征可以反映腫瘤負擔和亞型。但在大規模臨床隊列中仍然缺乏mCRPC的全基因組cfDNA甲基化研究。最近,細胞游離甲基化DNA免疫沉淀與下一代測序(cfMeDIP-seq)的發展為分析cfDNA甲基化組提供了一種有效的方法。這種方法可以從微量cfDNA中敏感地檢測ctDNA,并且與全基因組亞硫酸鹽測序方法相比更具成本效益。
近日,廈門大學健康醫療大數據國家研究院吉國力教授團隊和加拿大多倫多大學瑪格麗特公主癌癥中心何厚勝(Housheng Hansen He)教授團隊等合作在Nature子刊《nature communications》發表題為“The cell-free DNA methylome captures distinctions between localized and metastatic prostate tumors”的科研成果,研究利用cfMeDIP-seq技術分析了60個原發和175個轉移性前列腺癌樣本的細胞游離DNA(cfDNA)甲基化組,全基因組甲基化組高效捕獲了反映疾病異質性變異。進一步研究表明,cfDNA甲基化組可以以高準確率區分不同的疾病狀態,突出其作為疾病監測和預后的微創策略潛力。

標題:The cell-free DNA methylome captures distinctions between localized and metastatic prostate tumors(cfDNA甲基化組捕獲區分原發和轉移性前列腺癌)
影響因子: IF 14.7 / 1區
技術平臺:cfMeDIP-seq
作者利用高度敏感的富集型測序技術cfMeDIP-seq,分別對4個研究隊列中60名原發前列腺癌患者和175名轉移性前列腺癌患者的cfDNA甲基化組進行了分析。分析結果揭示cfDNA甲基化組可以捕獲腫瘤變化。在轉移性樣本中觀察到全基因組高甲基化,同時著絲粒周圍區域出現低甲基化。糖皮質激素受體(glucocorticoid receptor,GR)基因NR3C1的啟動子區域高甲基化與免疫信號減少有關。cfDNA甲基化組反映了臨床結果,并且可以以98.9%高精度準確率預測區分不同的疾病類型(轉移性和原發樣本),表現出了強大的疾病監測和預測的微創策略潛力。最后,研究從數據中預測拷貝數變化的能力,為有限的生物樣本提供了聯合遺傳和表觀遺傳分析的機會。
研究設計
樣本收集:4個研究隊列(原發性腫瘤患者來自CPC-Gene,轉移性前列腺癌來自VPC、Barrier、WCDT)。分析方法:使用高靈敏度的基于富集測序技術cfMeDIP-seq,分析了60個局部前列腺癌和175個轉移性前列腺癌患者的血漿DNA甲基化組,將所有血漿樣本中的甲基化DNA片段沉淀并進行配對末端DNA測序。
結果圖形
(1)對原發和轉移性前列腺癌(PCa)的血漿游離DNA甲基化組進行全基因組范圍分析。

圖1:cfMeDIP數據捕獲原發與轉移性去勢抵抗性前列腺癌(mCRPC)患者血漿樣本中cfDNA甲基化和片段化變化。
B-D. WCDT隊列,組織WGBS和cfMeDIP數據的樣本配對相關性,比較匹配樣本與非匹配樣本的差異。
E. %ctDNA、LDH以及與不依賴%ctDNA的LDH解釋的不同基因組位點富集情況。
F. 前10,000個變異區間的主成分分析(PCA)對四個隊列樣本進行三維表示。
G. 各隊列中較長cfDNA片段比例。
H. VPC隊列中mCRPC樣本的較長片段比例與ctDNA比例(%ctDNA)的皮爾遜相關性。
I-J. 片段大小與總生存期(OS)和無進展生存期(PFS)的相關性。
K. 樣本內片段比例標準差分布。
(2)cfDNA甲基化分析顯示轉移性前列腺癌樣本中廣泛高甲基化和著絲粒周圍區域低甲基化

圖2:cfMeDIP揭示轉移樣本中廣泛的高甲基化和著絲粒區域低甲基化。
A. 通過比較轉移性和原發前列腺癌,鑒定差異性甲基化區域(DMRs)火山圖。分別鑒定出2493個低甲基化和19048個高甲基化DMRs。B-C. 高甲基化(Hyper)和低甲基化(Hypo)DMRs平均甲基化水平與%ctDNA之間的皮爾遜相關性。
D-E. 高甲基化和低甲基化DMRs的基因組分布。
F. 與低甲基化峰重疊的重復類型分布的列聯表。
G. 與下調峰重疊的重復類型頻率。
H. 位于著絲粒周圍1 Mb區域內的差異性甲基化峰。
I. 染色體2中著絲粒附近區域信號分布示例。
J-K. 分別展示了cfMeDIP-seq和WGBS數據中H中顯示的差異峰的平均甲基化水平。
(3)NR3C1啟動子甲基化水平與不同疾病結局相關

圖3:GR基因甲基化差異與mCRPC改變相關。
A. GR基因(NR2C1)及其不同異構體示意圖。藍色條表示已鑒定的異常DMR。B. VPC隊列中GR位點甲基化水平與%ctDNA之間的皮爾遜相關性。
C-E. 癌癥基因組圖譜(TCGA)、中國前列腺癌基因組和表觀基因組圖譜(CPGEA)和WCDT隊列中GR位點甲基化與疾病結果之間的相關性。
F-H. TCGA(F)、CPGEA(G)和WCDT(H)隊列中高GR-DMR甲基化組中下調基因的生物學過程(BP)的基因本體(GO)富集分析。
(4)cfDNA甲基化組可高精度區分轉移性和原發性樣本

圖4:甲基化譜以高準確率區分原發和轉移性樣本。
A. 使用甲基化配置文件構建隨機森林預測因子工作流程。B. 測試數據集上,基于甲基化譜預測因子的預測準確性和接收者操作特征曲線下面積(AUROC)。VPC-V驗證樣本來自VPC隊列,OHS為安大略省健康研究隊列,DMR為差異甲基化區域。
C. 對于轉移樣本,正確或錯誤分類的樣本中%ctDNA分布。
(5)游離DNA甲基化組可以預測大規模遺傳變異

圖5:使用cfDNA甲基化組預測樣本拷貝數變異(CNA)。
B. VPC隊列中mCRPC樣本預測的CNA覆蓋度與%ctDNA之間的相關性。
C. 比較先前panel測序獲得的標準狀態和cfMeDIP-seq數據預測結果的基因CNA,針對CNA覆蓋度最高的前10個樣本。

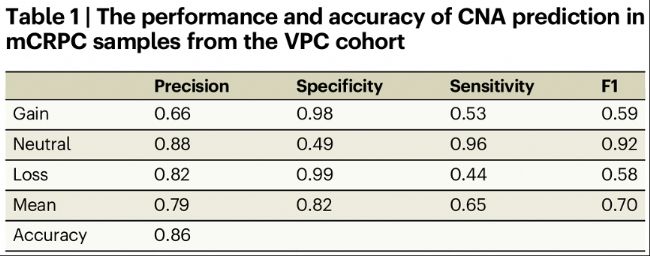
表1:VPC隊列中mCRPC樣本中CNA預測的性能和準確性
參考文獻:
Chen S, et al. The cell-free DNA methylome captures distinctions between localized and metastatic prostate tumors. Nat Commun. 2022 Oct 29;13(1):6467. pii: 10.1038/s41467-022-34012-2. doi: 10.1038/s41467-022-34012-2. PubMed PMID: 36309516.
標簽:
DNA甲基化
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com